NaN(Not a Number), NULL 과 같은 값들이 훈련 샘플에 오류로 발생하기 쉽다.
이와 같은 누락된 값들은 단순히 무시했을 때, 예상치 못한 결과를 만들어 낼 수 있다.
따라서 분석을 진행하기 전에 누락된 값을 처리하는 것이 중요하다.
테스트를 위한 CSV 예제 데이터셋
import pandas as pd
from io import StringIO
csv_data=\
'''A,B,C,D
1.0,2.0,3.0,4.0
5.0,6.0,,8.0
10.0,11.0,12.0,'''
df=pd.read_csv(StringIO(csv_data))
df
A B C D
0 1.0 2.0 3.0 4.0
1 5.0 6.0 NaN 8.0
2 10.0 11.0 12.0 NaN
A 0
B 0
C 1
D 1
dtype: int64
scikit-learn API 는 넘파이 배열 처리가 더욱 성숙하기 때문에 넘파일 배열을 사용하는 것이 더욱 좋음
df.values를 통해 dataframe을 numpy 배열로 바꿀 수 있다.
array([[ 1., 2., 3., 4.],
[ 5., 6., nan, 8.],
[10., 11., 12., nan]])
누락된 데이터 처리 방법
dropna()
dropna를 통해서 해당 훈련 샘플(행)이나 특성(열)을 완전히 삭제하는 방법
axis default value is 0
inplace 매개변수를 기본값 False 에서 True로 바꾸면, 새로운 데이터프레임을 반환하지 않고
주어진 데이터프레임 자체를 바꾼다.
A B C D
0 1.0 2.0 3.0 4.0
A B
0 1.0 2.0
1 5.0 6.0
2 10.0 11.0
A B C D
0 1.0 2.0 3.0 4.0
1 5.0 6.0 NaN 8.0
2 10.0 11.0 12.0 NaN
A B C D
0 1.0 2.0 3.0 4.0
A B C D
0 1.0 2.0 3.0 4.0
2 10.0 11.0 12.0 NaN
누락된 데이터를 제거하는 것은 간단해 보이지만 너무 많은 데이터를 제거하면
안정된 분석이 불가능 하게 된다. 또는 너무 많은 특성 열을 제거하면 분류기가
클래스를 구분하는데 필요한 중요한 정보를 잃을 위험이 있다.
interpolation(보간)
특성열을 통째로 제거하기 어려울 때 보간 기법을 사용하면, 데이터셋에 있는 다른 훈련 샘플로부터
누락된 값을 추정할 수 있다. 가장 대표적인 방법이 평균으로 대체하는 방법이다.
from sklearn.impute import SimpleImputer
import numpy as np
imr=SimpleImputer(missing_values=np.nan, strategy='mean')
imr=imr.fit(df.values)
imputed_data=imr.transform(df.values)
imputed_data
array([[ 1. , 2. , 3. , 4. ],
[ 5. , 6. , 7.5, 8. ],
[10. , 11. , 12. , 6. ]])
이외에 strategy로 median, most_frequent, constant 를 사용할 수 있다.(기본값 mean)
missing_values default value is np.nan
FunctionTransformer
SimpleImputer에는 axis 매개변수가 따로 없다. 기본 동작방식은 Imputer 클래스의 axis=0일 때와 같다.
행 방향으로 대체할 값을 차기 위해서는 FunctionTransformer를 사용하여 처리하여야 한다.
from sklearn.preprocessing import FunctionTransformer
ftr_imr=FunctionTransformer(lambda X:imr.fit_transform(X.T).T, validate=False)
imputed_data=ftr_imr.fit_transform(df.values)
imputed_data
array([[ 1. , 2. , 3. , 4. ],
[ 5. , 6. , 6.33333333, 8. ],
[10. , 11. , 12. , 11. ]])
SimleImputer 클래스의 add_indicator 매개변수를 True로 지정하면 indicator_ 속성이 추가되고
transform 메서드가 누락된 값의 위치를 포함된 배열을 반환
imr=SimpleImputer(add_indicator=True)
imputed_data=imr.fit_transform(df.values)
imputed_data
array([[ 1. , 2. , 3. , 4. , 0. , 0. ],
[ 5. , 6. , 7.5, 8. , 1. , 0. ],
[10. , 11. , 12. , 6. , 0. , 1. ]])
indicator_ 속성은 Missingindicator 클래스의 객체로 MissingIndicator 객체의 features_ 속성은 누락된
값이 있는 특성의 인덱스를 담고 있다.
imr.indicator_.fit_transform(df.values)
array([[False, False],
[ True, False],
[False, True]])
fit_transform 메서드를 호출하면, features_ 속성에 담긴 특성에서 누락된 값의 위치를 나타내는 배열을 반환
imr.inverse_transform(imputed_data)
array([[ 1., 2., 3., 4.],
[ 5., 6., nan, 8.],
[10., 11., 12., nan]])
SimpleImputer는 한 특성의 통계 값을 사용하여 누락된 값을 채운다.이와 달리 IterativeImputer 클래스는 다른 특성을 사용하여 누락된 값을 예측한다.
initial_strategy 매개변수에 지정된 방식으로 누락된 값을 초기화한다.
누락된 값이 있는 한 특성을 타깃으로 삼고 다른 특성을 사용해서 모델을 훈련하여 예측한다.
누락된 값이 있는 모든 특성을 순회한다.
initial_strategy 매개변수에 지정할 수 있는 값은 동일하게 mean, median, most_frequent, constant가 가능하다.
예측할 특성을 선택하는 순서는 적은 특성부터 선택하는 ascending, 누락된 값이 가장 큰 특성부터 선택하는 descending
왼쪽에서 오른쪽으로 선택하는 roman, 오른쪽에서 왼쪽으로 선택하는 arabic 랜덤하게 고르는 random이 있다.
default값은 ascending이다.
특성 예측은 종료 조건을 만족할 때까지 반복한다.
각 반복 단계에서 이전 단계와 절댓값 차이 중 가장 큰 값이 누락된 값을 제외하고
가장 큰 절대값에 tol 매개변수를 곱한 것보다 작을 경우 종료한다. tol 매개변수의 기본값은 1e-3이다.
max_iter default값은 10이다.
예측에 사용하는 모델은 estimator 매개변수에서 지정할 수 있으며, 기본적으로 BayesianRidge 클래스를 사용한다.
예측에 사용할 특성 개수는 n_nearest_features에서 지정할 수 있으며, 상관 계수가 높은 특성을 우선하여
랜던하게 선택된다. 기본값은 None으로 모든 특성을 사용한다.
IterativeImputer 클래스는 아직 실험적이기 때문에 enable_iterative_imputer 모듈을 임포트해야 한다.
from sklearn.experimental import enable_iterative_imputer
from sklearn.impute import IterativeImputer
iimr=IterativeImputer()
iimr.fit_transform(df.values)
array([[ 1. , 2. , 3. , 4. ],
[ 5. , 6. , 7.00047063, 8. ],
[10. , 11. , 12. , 12.99964527]])
KNNImputer 클래스는 k-최근접 이웃 방법을 사용하여 누락된 값을 채운다.
n_neighbors 매개변수로 최근접 이웃의 개수를 지정하며 기본값은 5이다.
샘플 개수가 n_neighbors보다 작으면, SimpleImputer(strategy=‘mean’)과 결과가 같다.
from sklearn.impute import KNNImputer
kimr=KNNImputer()
kimr.fit_transform(df.values)
array([[ 1. , 2. , 3. , 4. ],
[ 5. , 6. , 7.5, 8. ],
[10. , 11. , 12. , 6. ]])
fillna()
A B C D
0 1.0 2.0 3.0 4.0
1 5.0 6.0 7.5 8.0
2 10.0 11.0 12.0 6.0
df.fillna(method='bfill')
A B C D
0 1.0 2.0 3.0 4.0
1 5.0 6.0 12.0 8.0
2 10.0 11.0 12.0 NaN
df.fillna(method='ffill')
A B C D
0 1.0 2.0 3.0 4.0
1 5.0 6.0 3.0 8.0
2 10.0 11.0 12.0 8.0
df.fillna(method='ffill', axis=1)
A B C D
0 1.0 2.0 3.0 4.0
1 5.0 6.0 6.0 8.0
2 10.0 11.0 12.0 12.0
axis=1로 지정해주면 행이 아니라, 열을 사용한다.
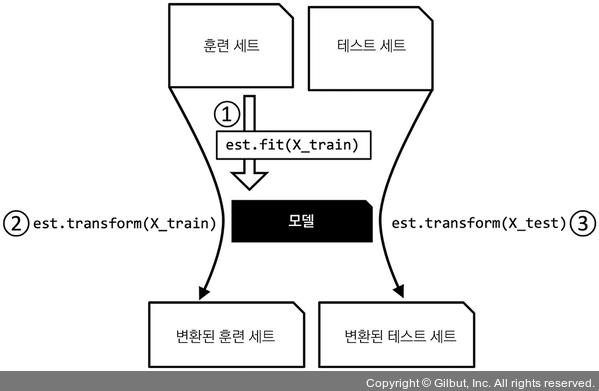
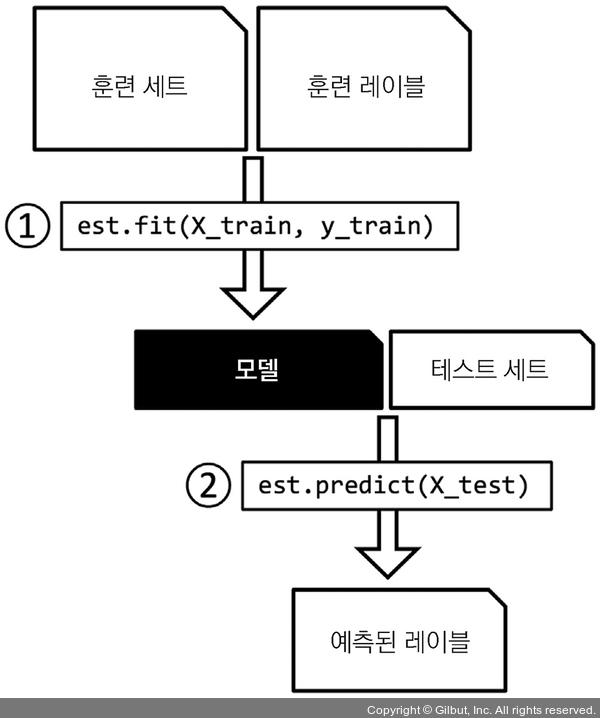
SimpleImputer 클래스는 데이터 변환에 사용되는 사이킷런의 변환기(transformer) 클래스이다.
주요 메서드는 fit와 transform 이다.
fit 메서드를 이용하여 훈련데이터에서 모델 파라미터를 학습한다.
transform 메서드를 사용하여 학습한 파라미터로 데이터를 변환한다.
(변환할려는 데이터 배열은 모델 학습에 사용한 데이터의 특성 개수와 같아야 한다.)
추정기는 predict 메서드가 있지만, transform 메서드도 가질 수 있다.
predict 메서드를 사용하여, 레이블이 없는 새로운 데이터 샘플에 대한 예측을 만든다.